有了RNA-protein 相互作用的研究方法,再也不愁高分的SCI文章了!
熱門研究的非編碼RNA,包含LncRNAs、snoRNAs和mRNA UTR,其許多功能是與RNA結合蛋白(RBPs)直接相互作用實現的。最近研究發現數百種新RBPs的RNA結合域尚不清楚,于是強調RNA蛋白復合物的復雜性和多樣性,進而最新研究成果擴展了RNA-protein 相互作用的兩大類方法:一類方法是以RNA為中心,研究與感興趣的RNA結合的protein;另一類方法是以蛋白質為中心,研究與感興趣的protein結合的RNAs。由于每種方法都有其優勢和局限,選擇最佳方法來處理生物學問題變得非常重要。(Review,Methods to study RNA-protein interactions,IF=28.464)
一、以RNA為中心的方法:發現與感興趣的RNA結合的RBPs
1.以RNA為中心的方法示意圖
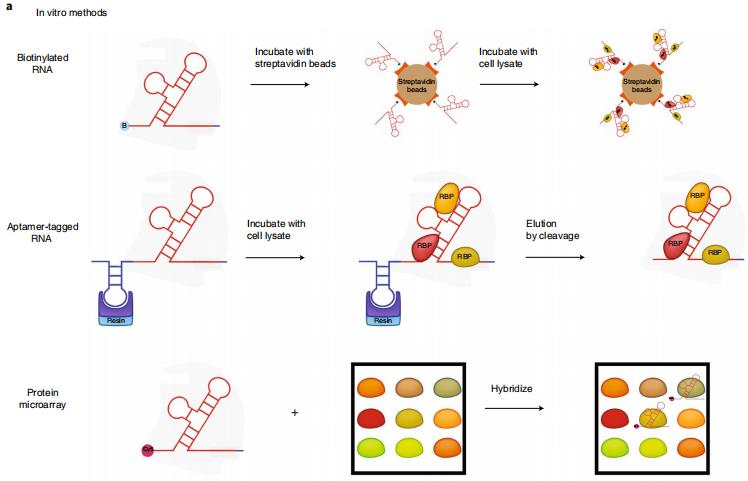
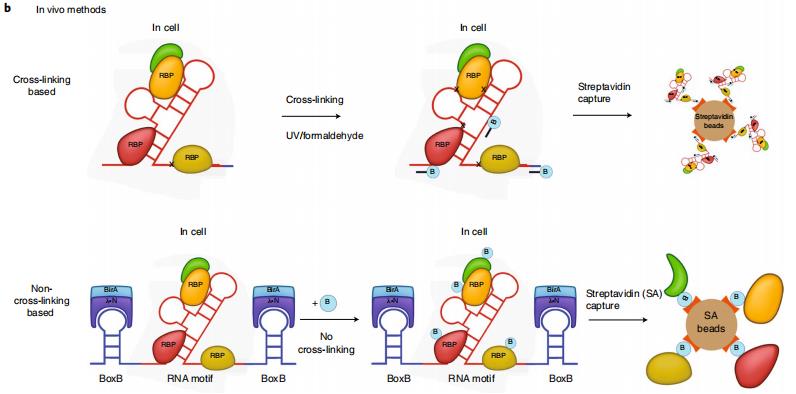
以RNA為中心的方法又分體內方法和體外方法。
體外方法:
方法a(1)是用于pull down 實驗的RNA 5 '或3 ' RNA生物素化。末端生物素化RNA與streptavidin beads(鏈霉親和素珠)結合,然后加入細胞提取物孵育后洗滌。此方法不需要洗脫RNA,向beads加入SDS,煮沸,用于蛋白質組學分析。
方法a(2)是感興趣的RNA加適體標簽,例如,S1適體標簽與streptavidin beads結合,生物素可以競爭性地結合streptavidin ,以洗脫標記有S1適體的RNA。已有文獻報導,Cys4 核糖核酸內切酶可以結合Cys4 hairpin loop,用imidazole(咪唑)將核酸酶從發夾環上分離,釋放結合RNA的RBPs。優缺點:降低了非特異性的結果。
方法a(3)標記Cy5染料的體外轉錄(IVT )RNA雜交到約9400個重組人蛋白(Human ProtoArray)的蛋白芯片。通過熒光檢測結合Cy5 RNA的蛋白質。優缺點:Human ProtoArray方法不需要細胞提取物,有助于發現RNA-protein 相互作用的一種研究方法。然而,這類方法限制于斑點蛋白的折疊結構和翻譯后修飾以及濃度的改變。
體內方法:
甲醛是一種小的雙功能交聯劑,易滲透細胞并在2個單位內交聯大分子,包括蛋白質-蛋白質、蛋白質-DNA和蛋白質-RNA復合物交聯,這種交聯是可逆的。UV light 交聯是一種零距離交聯且不可逆。與甲醛交聯劑相比,UV light 不交聯蛋白質-蛋白質相互作用,特異性強,但效率較低。
方法b(1)是通過蛋白-RNA交聯進而鑒定體內的相互作用。具體內容是在變性條件下去除非共價相互作用來純化RNA,生物素化寡核苷酸探針與感興趣的RNA雜交, RNA和交聯蛋白被純化與鑒定。優缺點:效率低,細胞用量多。
方法b(2)RNA-蛋白質相互作用檢測法簡稱RaPID (LN-HA-Bira),BoxB RNA莖環側面結合感興趣RNA,在含有生物素的培養基培養細胞,BoxB位點結合融合蛋白(λ-N ),融合蛋白引起與感興趣RNA結合的蛋白生物素化,最后用streptavidin beads捕獲生物素化的蛋白質。
| 方法 | 應用 | 優點 | 缺點 |
| RNA生物素化 | SMN mRNA | RNA末端標記生物素,與鏈霉親和素珠結合強 | 在體外,可能偏好細胞提取物中豐富的蛋白質 |
| S1適體 | ARE motif | 用生物素易洗脫RBP復合物從streptavidin beads | 在體外,可能偏好豐富的蛋白質 |
| Cys4 | Pre-miRNA | 用咪唑洗脫RBP復合物 | 在體外,可能偏好豐富的蛋白質 |
| 蛋白芯片 | TINCR, SNORD50 | 不需要細胞提取物;不需要MS | 在體外,僅與微陣列上的蛋白質直接相互作用 |
| RAP | Xist, FIRRE noncoding RNA | 體內;運用UV交聯和長的寡核酸探針(120nt),特異性高 | 耗費細胞量大 |
| TRIP | p27 mRNA, CEP-1 mRNA | 體內;紫外交聯特異性高。 | Poly(A)和生物素化ASO的捕獲使效率降低 |
| PAIR | ANK mRNA | 體內;紫外交聯特異性高。 | 肽核酸生產耗費成本和精力 |
| MS2-BioTRAP | IRES | 體內;紫外交聯特異性高。 | 需要MS2與RNA的結合,RNA和標簽蛋白的轉染/感染,耗費細胞量大 |
| CHART | Xist, MALAT1, NEAT1 | 體內 | 需要RNase識別探針位置的;耗費細胞量大 |
| ChIRP | TERC, Xist | 體內,探針設計無需要特異性RNA | 短探針可能會拉下類似的片段序列;耗費細胞量大 |
| RaPID | ZIKV-宿主蛋白相互作用 3’端UTR Motifs | 體內;所需細胞數量少;直接標記蛋白質 | 需要BoxB與RNA連接;限制短序列;RNA和標簽蛋白的轉染/感染 |
二、以蛋白為中心的方法:發現與感興趣的蛋白質結合的RNAs
1.蛋白質純化的方法
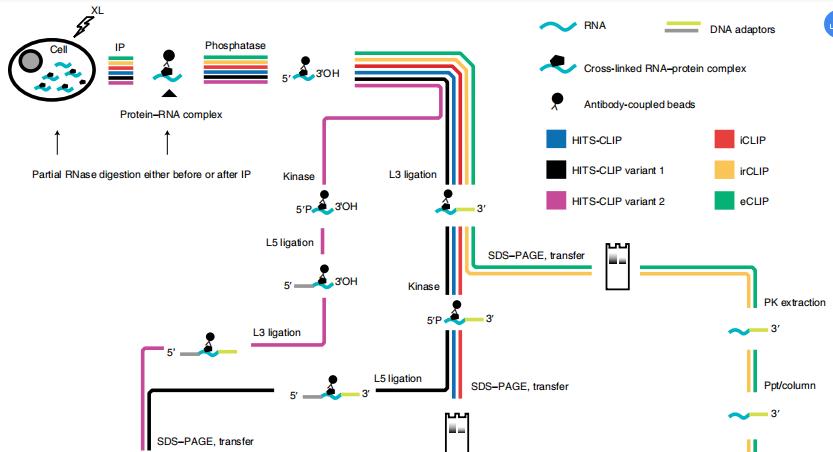
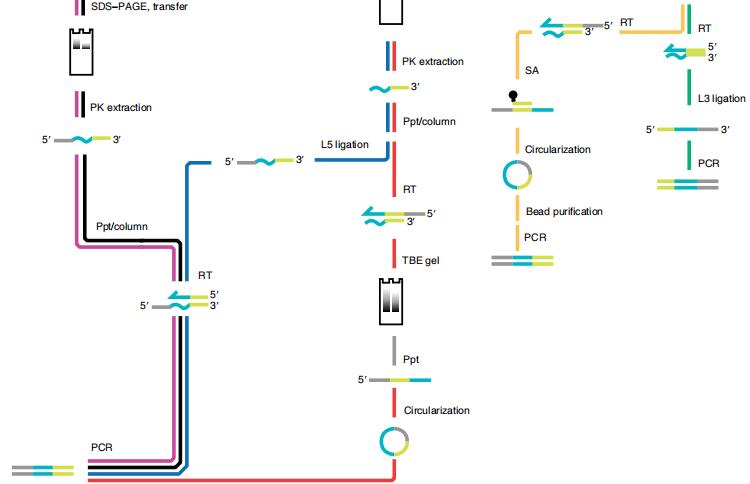
紫外交聯免疫沉淀結合高通量測序(CLIP-seq)原理
基于RNA分子與RNA結合蛋白在紫外照射下發生耦聯,以RNA結合蛋白的特異性抗體將RNA-蛋白質復合體沉淀之后,回收其中的RNA片段,經添加接頭、RT-PCR等步驟,對這些分子進行高通量測序,再經生物信息學的分析和處理、總結,挖掘出其特定規律,在體內與眾多 RNA 靶標的結合模式,并揭示其在一些重要生物學過程中的功能,是一項在全基因組水平揭示 RNA分子與RBP相互作用的革命性技術。能夠確定RBP與RNA直接的相互作用和精確的結合位點。
CLIP-seq 的應用范圍:
(1)繪制全基因組范圍的RNA和RBP相互作用圖譜,解析“Argonaute(Ago)—miRNA—mRNA”三者的相互作用;
(2)RBP與miRNA、lncRNA等非編碼RNA的作用網絡與功能的發現;
(3)與 RIP-seq 相比,可以鑒定RBP和RNA之間的直接相互作用,確定 RBP 與miRNA、lncRNA 等非編碼 RNA 的精確結合位點。
上圖展示了從免疫沉淀到PCR,該圖顯示CLIP-seq的一些重要步驟。例如:XL,UV交聯;IP,免疫反應;磷酸酶,3’端去磷酸化;激酶,5’端磷酸化;RT,逆轉錄;L3,RNA或DNA 3’端適體連接;L5,5’端適體連接;PK提取,從硝基纖維素膜中提取蛋白酶K;Ppt/柱,乙醇沉淀或柱分離核酸;TBE、Tris-B酸鹽-EDTA;SA、抗生物素蛋白。
2.不需要蛋白質純化的方法
在不純化RBP的情況下識別RBP的靶RNA的方法是相對較新的,目前主要是對RNA進行兩種不同化學修飾。第一種是TRIBE,感興趣的蛋白質融合到ADAR酶,在ADAR使附近的腺嘌呤脫氨后,通過測序鑒定脫氨堿基。第二種是RNA標記法,poly(U)多聚酶與感興趣的RBP融合后,添加poly(U)尾到結合的RNA。RNA 3’末端序列識別poly(U)尾;在特定亞細胞區過氧化物酶標記RBP,用于識別結合的RNA,這種方法還沒有被用來鑒定靶RNA。酶修飾RNA的方法是可行的,以后會發現更多的酶修飾RNA。
我們希望更多的技術在這一領域快速發展,多種差異的化學標記分離RBP對于RBP純化組合調控和化學修飾組合研究,以此研究RBP-RNA相互作用的位置。






