scRNA-seq和scATAC-seq聯合分析揭示ccRCC異質性和調控動態
透明細胞腎細胞癌(ccRCC)經常具有高度的腫瘤異質性。在單細胞水平上闡明ccRCC的染色質圖譜可以加深對該疾病的功能狀態和調控動態的理解。在這里,作者對19個ccRCC樣本進行了單細胞RNA測序(scRNA-seq)和單細胞轉座酶可及染色質測序(scATAC-seq),并利用全外顯子組測序來了解個體間的異質性。構建了ccRCC的單細胞轉錄組和染色質可及性圖譜,以揭示ccRCC中不同腫瘤細胞亞型的調控特征。研究發現了兩個促進ccRCC侵襲和遷移的長非編碼RNA(RP11-661C8.2和CTB-164N12.1),并通過體外實驗進行了驗證。綜上所述,本研究全面描述了ccRCC的基因表達和DNA調控圖譜,可為ccRCC的生物學和治療提供新的見解。本文于2023年3月發表在《Cancer Research》IF:11.2期刊上。
技術路線:

主要實驗內容:
1、單細胞多組學研究描繪了人類ccRCC轉錄組景觀
考慮到先前在ccRCC進行的單細胞轉錄組研究數量,作者希望通過單細胞多組學方案(圖1A)進一步發現表觀遺傳調控機制。從19名患有局部ccRCC的患者中收集了新鮮的手術切除物制備單細胞懸液,一部分用于scRNA-seq,而其余部分則用于scATAC-seq(圖1A)。額外的組織或單細胞懸液用于大規模ATAC-seq或WES,如圖1A所示。經過Seurat質控后,從ccRCC中捕獲到了102,723條高質量的單細胞轉錄組信息。為減輕批次效應,使用Harmony跨樣本匯總細胞并聚類。基于先前研究中標記基因的表達和細胞注釋,將ccRCC分類為22個獨立的細胞類型,包括四種腫瘤細胞亞型(ccRCC 1、ccRCC 2、ccRCC 3和ccRCC 4)、四種內皮細胞亞型(Endo cells 1、Endo cells 2、Endo cells 3和Endo cells 4)等(圖1B和C)。在ccRCC中,發現T細胞(30,339,29.53%)最多,其次是腫瘤細胞(19,819,19.29%;圖1D)。由于ccRCC的腫瘤異質性,對來自19名不同個體的腫瘤樣本進行了scRNA-seq。發現大多數細胞類型都來自每個樣本,而腫瘤細胞和內皮細胞之間的個體差異更為顯著(圖1E)。例如,腫瘤細胞(ccRCC 4)僅來源于患者RCC84,而Endo cells 4僅來自三名個體(圖1E)。與先前研究一致,免疫細胞的廣泛浸潤,特別是T細胞和TAMs,表征了ccRCC的免疫微環境(圖1B和D)。
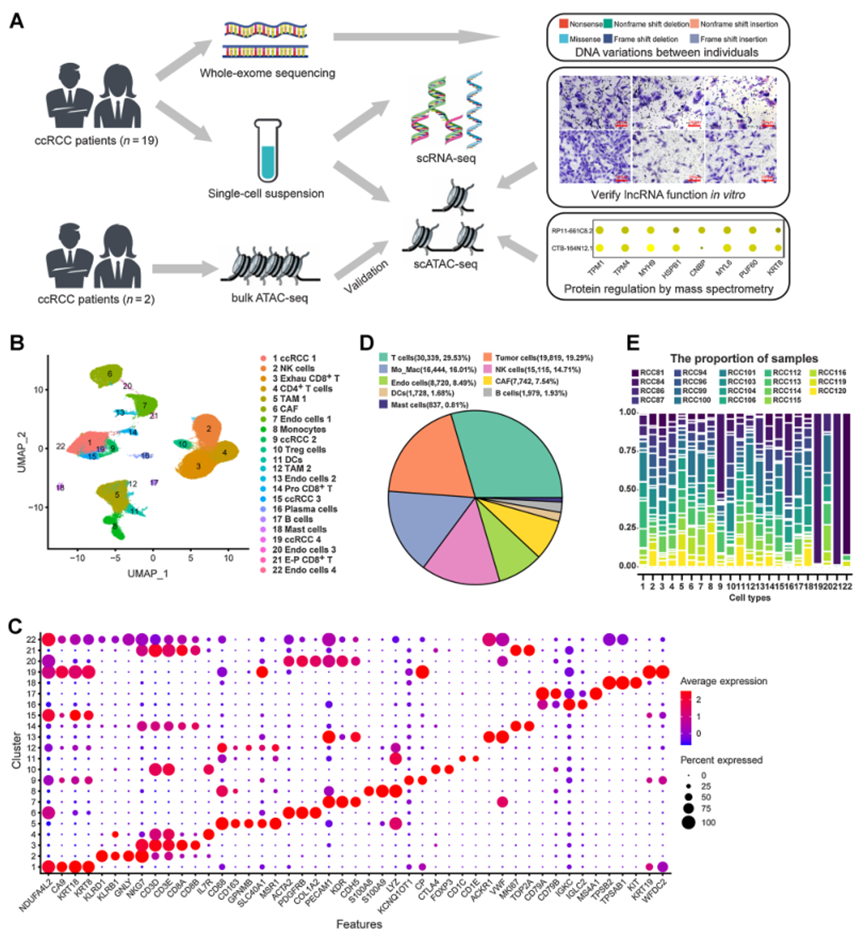
圖1 ccRCC樣本的單細胞轉錄組圖譜總覽
2、通過scRNA-seq對腫瘤細胞的分子亞型進行表征
考慮到ccRCC的遺傳傾向,對所有ccRCC樣本進行了全外顯子測序以了解個體之間的DNA變異。發現這些常見突變基因的變異與先前研究中的相似(圖2A)。VHL是最常見的突變基因,其突變率在本研究中為84.2%(圖2A)。有趣的是,發現CA9(15.8%)、KRT14(15.8%)和CD24(47.3%)的突變率也較高,這些突變是錯義突變(圖2A)。將腫瘤細胞分為VHL、CA9、KRT14和CD24突變或非突變組,并比較它們的基因表達差異(圖2B–E)。發現突變和非突變的腫瘤細胞基因表達非常相似,皮爾遜相關系數大于0.98(圖2B–E)。因此,這也表明在ccRCC中的單一突變基因并沒有引起腫瘤細胞所有基因表達的巨大變化。
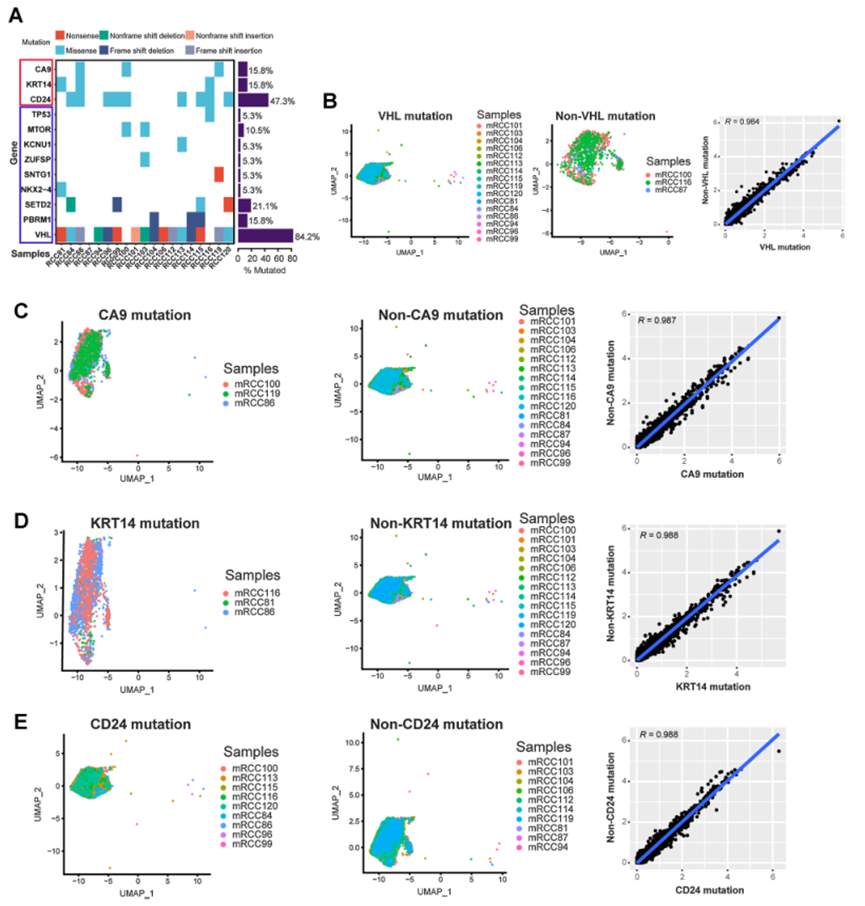
圖2 scRNA-seq揭示了ccRCC中基因突變與基因表達的關系
根據基因表達特征,腫瘤細胞通過scRNA-seq(圖3A)無偏地聚類成四個細胞亞型(ccRCC 1、ccRCC 2、ccRCC 3和ccRCC 4)。除ccRCC 4外,其余三個腫瘤細胞亞型都來自每個患者(圖3B)。盡管存在個體差異,但在腫瘤細胞中有對細胞亞群分類的良好代表性。最初,為客觀地揭示腫瘤細胞的特定基因表達,作者將其與鄰近正常上皮細胞進行比較,這些細胞來源于作者先前的研究。不同腫瘤細胞亞型中存在基因表達的顯著差異。作者發現ccRCC 1高表達CA9和NDUF4AL2,而ccRCC 2高表達KCNQ1OT1和CP(圖3C)。ccRCC 3高表達NDUF4AL2和CYB5A,而ccRCC 4表達腫瘤細胞中大多數高表達的基因(圖3C)。CA9和NDUF4AL2在先前的研究中已被確認為ccRCC的標記基因。CP在先前的單細胞研究中被確認為ccRCC的特異性標記物,這在本研究中從更大的ccRCC樣本中得到了證實。KCNQ1OT1是一個致癌lncRNA,在先前的研究中已有報道。然而,這是通過scRNA-seq首次發現KCNQ1OT1在ccRCC的特定細胞亞型中高表達。
根據scRNA-seq數據,進行拷貝數變異(CNV)分析,發現這些腫瘤細胞亞型存在不同的CNV特征(圖3D)。作者發現大多數腫瘤細胞的特征是染色體3(chr3)上的缺失和染色體5(chr5)上的增益,與TCGA對ccRCC的項目結果一致。這個結果在ccRCC 1中更為集中,而在ccRCC 4中不明顯(圖3D)。這可能是由于腫瘤細胞的克隆或亞克隆導致的。
擁有ccRCC的亞型信息后,作者試圖預測這四種腫瘤細胞類型的預后。先計算每個腫瘤細胞類型的差異表達基因(DEGs)。選擇每個細胞亞型的前100個DEGs,并基于在TCGA數據庫上對ccRCC的生存分析結果確定預后。發現ccRCC 1、ccRCC 2和ccRCC 3中的大多數DEGs表明積極的生存狀況,而ccRCC 4中的更多DEGs則表明不良預后,與前三個細胞亞型相比顯著(圖3E)。
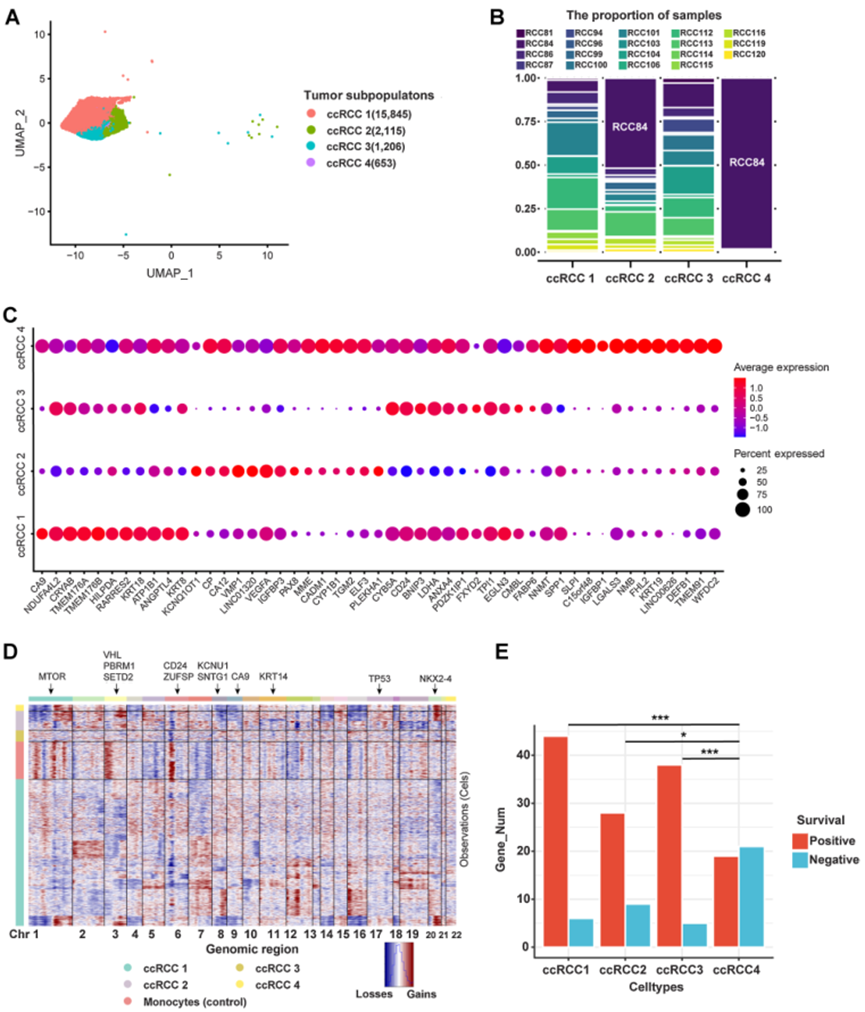
圖3 人ccRCC腫瘤細胞的異質性
3、構建ccRCC的單細胞染色質可及性景觀
在前一項研究中,已經構建了RCC中免疫細胞動態染色質景觀的單細胞圖譜,為理解ccRCC中免疫細胞的功能狀態和調控動態提供了豐富的資源。然而,需要構建一個更全面的ccRCC單細胞染色質可及性景觀,包括腫瘤細胞在內的大樣本,這可揭示腫瘤細胞的調控特征。在這項研究中,作者對19個ccRCC樣本進行了scATAC-seq。經過質控后,捕獲到了61,693個高質量細胞核和190,916個唯一峰值。將這些細胞分類為29種不同的細胞類型,并確定了每種細胞類型的樣本來源(圖4A和B)。scATAC-seq的細胞注釋計算了腫瘤細胞(CA9和KRT14)、內皮細胞(VWF和CDH5)、CAF(RGS5)、免疫細胞(PTPRC)、CD4+ T細胞(IL7R)、B細胞(SDC1)和NK細胞(KLRD1和KLRB1)的標記基因活性分數(圖4C)。隨后,對特定細胞類型的峰值進行了分析,然后確定了這些峰值的相應基因區域(圖4D)。基于以上工作,定義了ccRCC中的29種不同細胞類型(圖4A)。
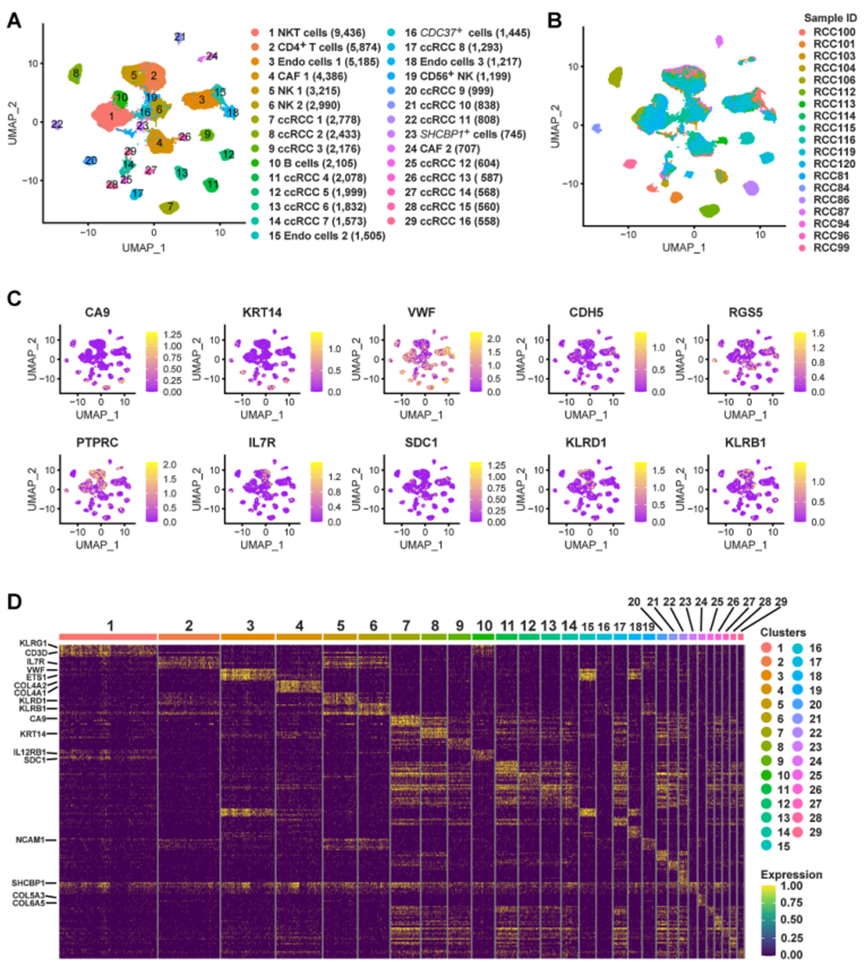
圖4 ccRCC的單細胞染色質可及性和外觀
利用scATAC-seq在所有細胞類型中確定了共同可及的染色質區域,例如在chr9和chr17中(圖5A)。這些共同可及的染色質區域還表明它們的調控過程并非細胞類型特異。因此,通過識別細胞類型特異的共同可及染色質區域,可以發現細胞類型特異的調控過程。此外,scATAC-seq結果顯示非腫瘤細胞(如免疫細胞、內皮細胞和CAF)的染色質可及性在個體間是一致的(圖5B)。每種細胞類型都具有良好的樣本普適性,并且幾乎來自所有樣本(圖5B)。然而,基于染色質可及性,腫瘤細胞可以被分類為16個細胞亞型(圖5C)。大多數腫瘤細胞亞型來自單個ccRCC樣本(圖5C)。類似于先前對基底細胞癌的研究,ccRCC中腫瘤細胞的染色質可及性顯示出顯著的個體間異質性。
4、scATAC-seq揭示ccRCC腫瘤細胞染色質調控的特點
為研究腫瘤細胞染色質調控的特征,利用scATAC-seq鑒定了16個腫瘤細胞亞型及每個亞型的染色質可及性區域(圖5C)。作者發現CA9和KRT14在ccRCC細胞的可及性區域中充當標記基因,它們在每個腫瘤細胞亞型中都保持染色質可及性(圖5D和E)。為更好地發現腫瘤細胞亞型之間的異質性,對這16個細胞亞型中的前10個染色質可及性區域進行富集,并將這些區域映射到基因上,發現這些染色質可及性區域既有重疊又有差異,呈現出一種不規則的狀態。這也可能表明ccRCC在DNA水平上存在遺傳傾向。
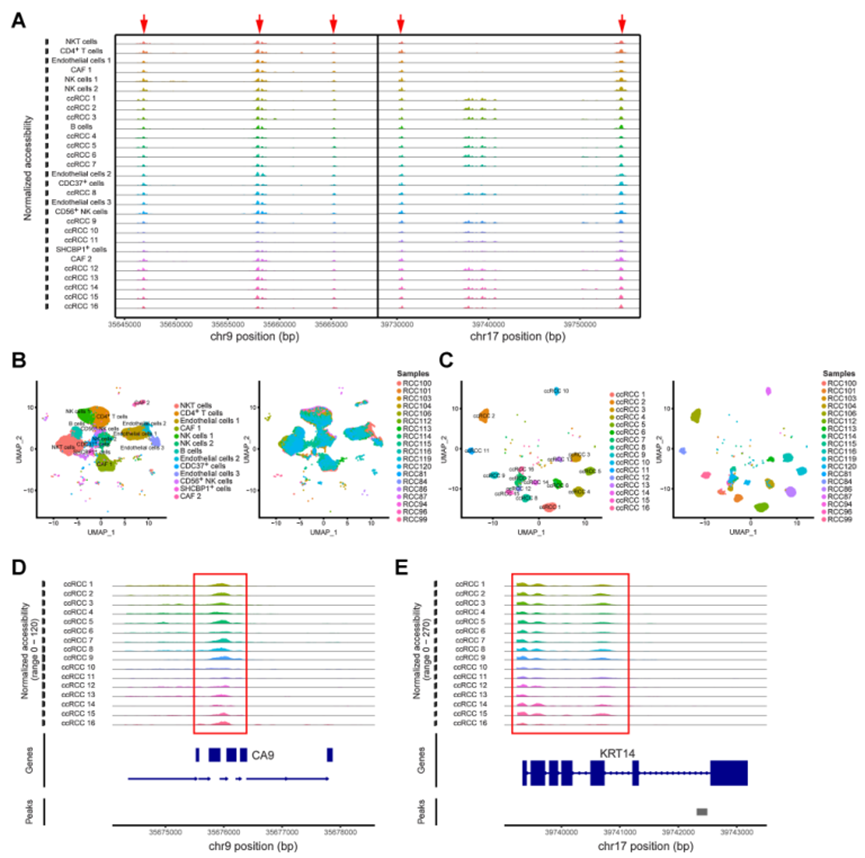
圖5 ccRCC單細胞染色質可及性特征
5、scATAC-seq揭示lncRNA在體外促進ccRCC侵襲和遷移
有趣的是,作者通過scATAC-seq發現了四種lncRNA(RP11-661C8.2、CTB-32H22.1、CTB-164N12.1和RP11-267A15.1),這些lncRNA在腫瘤細胞亞型中具有特異的可及性(圖6A)。所有這些lncRNA都有一個共同的特征,即位于染色體5上(圖6A)。研究證明cis-acting的lncRNA可以激活、抑制或以其他方式調節靶基因的表達。考慮到TCGA和本研究均證實ccRCC具有染色體5上拷貝數增加的特征,作者假設這些lncRNA可能在ccRCC中發揮特定的調控作用。因此,選擇了兩種lncRNA(RP11-661C8.2和CTB-164N12.1)進行實驗,分別利用ASO-5608和ASO-5717敲低 RP11-661C8.2和CTB-164N12.1。隨后驗證這些lncRNA的生物學功能。
發現經過ASO處理后,ccRCC細胞系的增殖活性顯著減少,包括786-O組和Caki-2組(圖6B)。CCK-8實驗證實ASO處理后ccRCC細胞系的增殖在24、48和72小時內均受到了抑制,與NC組相比(圖6C)。NC組的劃痕愈合優于ASO處理組,無論是在786-O還是Caki-2中(圖6D–F)。此外,Transwell實驗結果表明經過ASO處理后,786-O和Caki-2的遷移顯著減少(圖6G和H)。在ASO處理后,ccRCC細胞系中E-cadherin的表達與NC組相比顯著增加(圖6I)。這些結果確認RP11-661C8.2和CTB-164N12.1在體外促進ccRCC的侵襲和遷移。
作者通過質譜法鑒定了與RP11-661C8.2和CTB-164N12.1相互作用的蛋白質。根據可信的肽段和蛋白質,富集了與RP11-661C8.2和CTB-164N12.1密切相互作用的蛋白質,并展示最顯著的蛋白,如TPM1、TPM4、MYH9、HSPB1、CNBP、MYL6、PUF60和KRT8(圖6J)。發現這兩種lncRNA結合的蛋白質非常相似,除了CNBP更緊密地與RP11-661C8.2結合(圖6J)。隨后,將編碼這些蛋白質的基因放入scRNA-seq數據中,以估計它們在每個細胞亞型中的表達。有趣的是,這些基因在CAF和內皮細胞3中幾乎都高表達(圖6J)。一些基因,如TPM1、HSPB1和KRT8,在腫瘤細胞中高表達(圖6J)。因此,認為RP11-661C8.2和CTB-164N12.1的調控功能是細胞類型特異的,主要調控CAF、內皮細胞和腫瘤細胞。
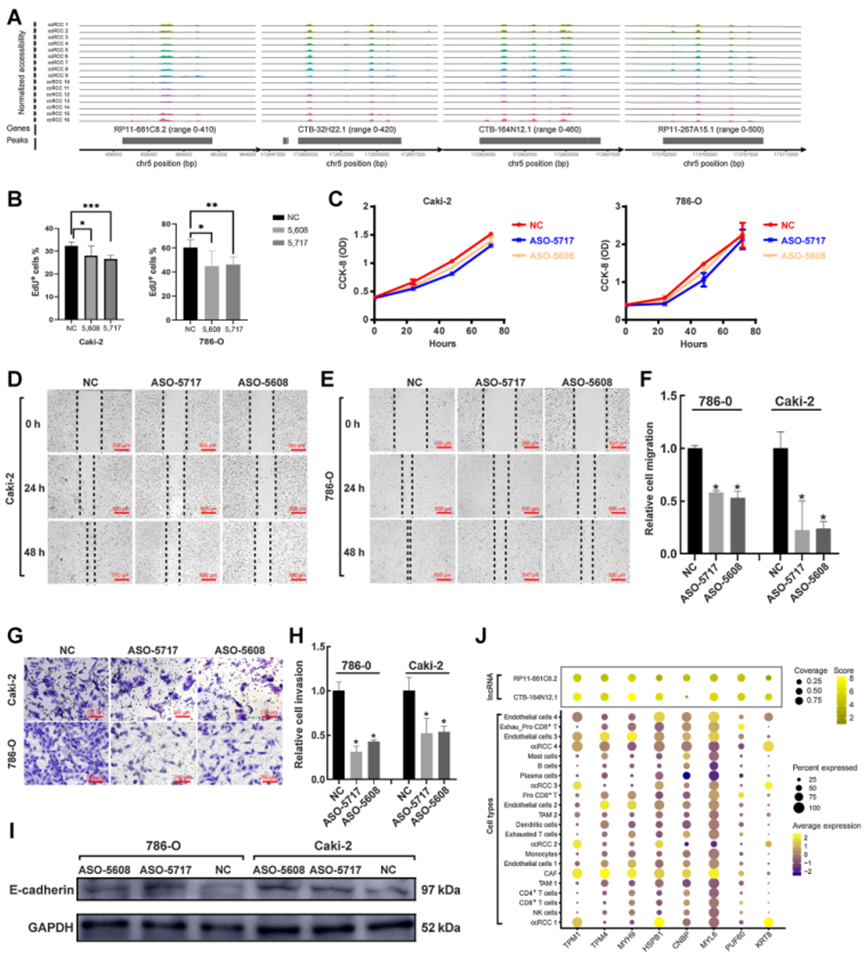
圖6 ccRCC腫瘤細胞中特異性lncRNA的發現和驗證
6、scATAC-seq揭示ccRCC的轉錄調控因子
scATAC-seq的另一個優勢是發現特定的調控過程,特別是轉錄因子(TFs)。由于這是單細胞研究,可以將這些TFs特異地定位到細胞類型。作者發現許多具有細胞類型特異性和TF結合基序的TFs。如SPIC和EOMES分別是B細胞和NK細胞中的特異性TFs(圖7A)。與先前的研究相比,基序的DNA堿基序列也非常相似(圖7A)。作者發現ZEB1充當了一個通用結合腫瘤細胞的TF,而ETV4是被非腫瘤細胞普遍結合的(圖7A)。此外,通過scATAC-seq鑒定了內皮細胞(SOX8)、CAFs(EBF2)和T細胞(ETS1)的特異性TFs和基序(圖7A)。通過富集每種細胞類型中前10個最顯著的TFs,發現這些TFs具有細胞類型特異性,尤其是在腫瘤細胞和免疫細胞之間(圖7B)。為預測這些細胞類型特異性TFs的確切結合位置,進行TF足跡分析(圖7C)。因此,正如前面提到的,定義scATAC-seq中的細胞類型時需要考慮TFs。
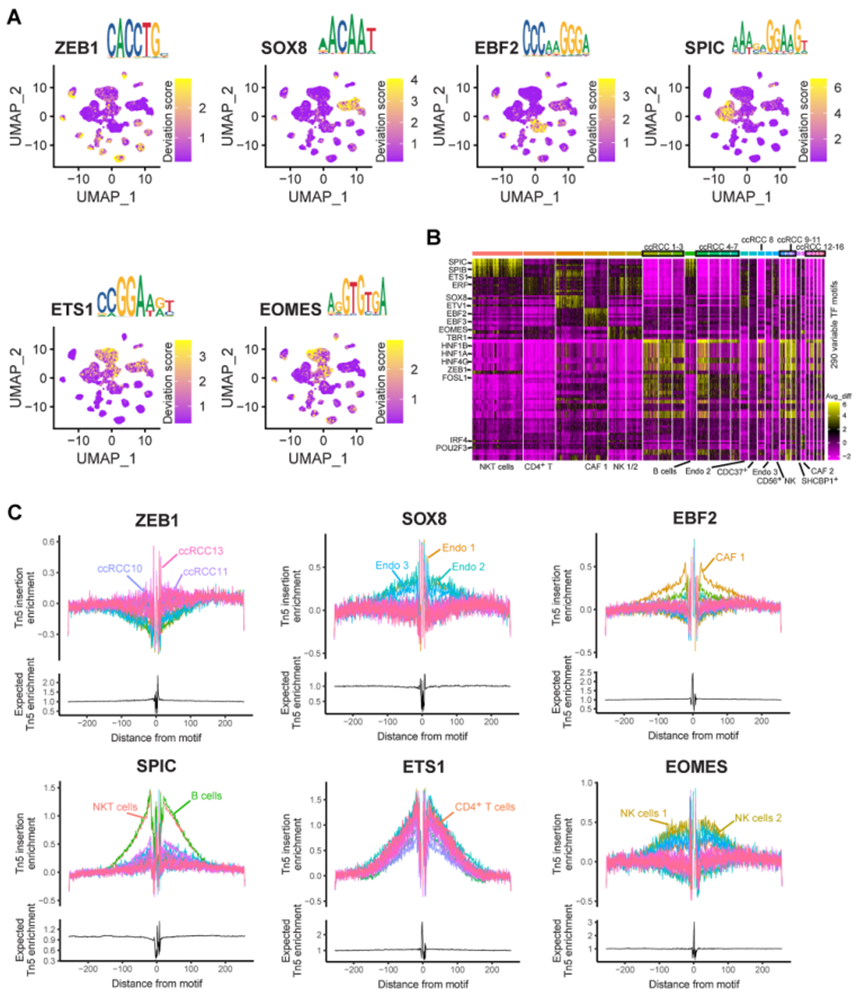
圖7 通過scATAC-seq鑒定ccRCC中TFs的特征
7、整合scATAC-seq和scRNA-seq分析揭示腫瘤細胞的調控特征
基因表達的調控是一個非常復雜的過程,受到表觀遺傳調控、lncRNA、增強子、啟動子等因素的影響。在這項研究中,作者通過整合scATAC-seq和scRNA-seq的結果,構建二者的共嵌圖發現ccRCC的調控規律(圖8A)。盡管通過空間投影反映的scATAC-seq和scRNA-seq數據存在差異,但相同細胞類型之間的分布非常相似(圖8A)。
通過整合scATAC-seq(16個亞型)和scRNA-seq(四個亞型)中的所有腫瘤細胞,發現腫瘤細胞亞型中染色質可及性與基因表達之間存在相關性(圖8B)。ccRCC 1(scRNA-seq亞型)的基因表達與15個腫瘤細胞類型(scATAC-seq亞型)的染色質可及性相關聯(圖8C),表明腫瘤細胞調控的復雜性。還發現一對腫瘤細胞類型,即ccRCC 2(scRNA-seq亞型)和ccRCC 11(scATAC-seq亞型),暗示染色質可及性與基因表達之間的對應關系(圖8C)。在先前的結果中,ccRCC 2和ccRCC 11主要來自樣本RCC84(圖2C)。因此,揭示由ccRCC 11的可及染色質區域調控的ccRCC 2的基因表達特征是準確的(圖8D)。然而,ccRCC 1(scRNA-seq亞型)的調控特征復雜,包括15個scATAC-seq腫瘤細胞亞型的調控特征(圖8E)。盡管如此,在所有15個腫瘤細胞亞型中發現了一些共同的可及染色質區域:“chr11-111906243-111914666”、“chr19-10352456-10354286”、“chr5-178286237-178290844”和“chr7-73829891-73835325”(圖8E)。這些發現表明,在研究的ccRCC腫瘤細胞的可及染色質區域存在個體差異,同時也存在共同的調控區域。
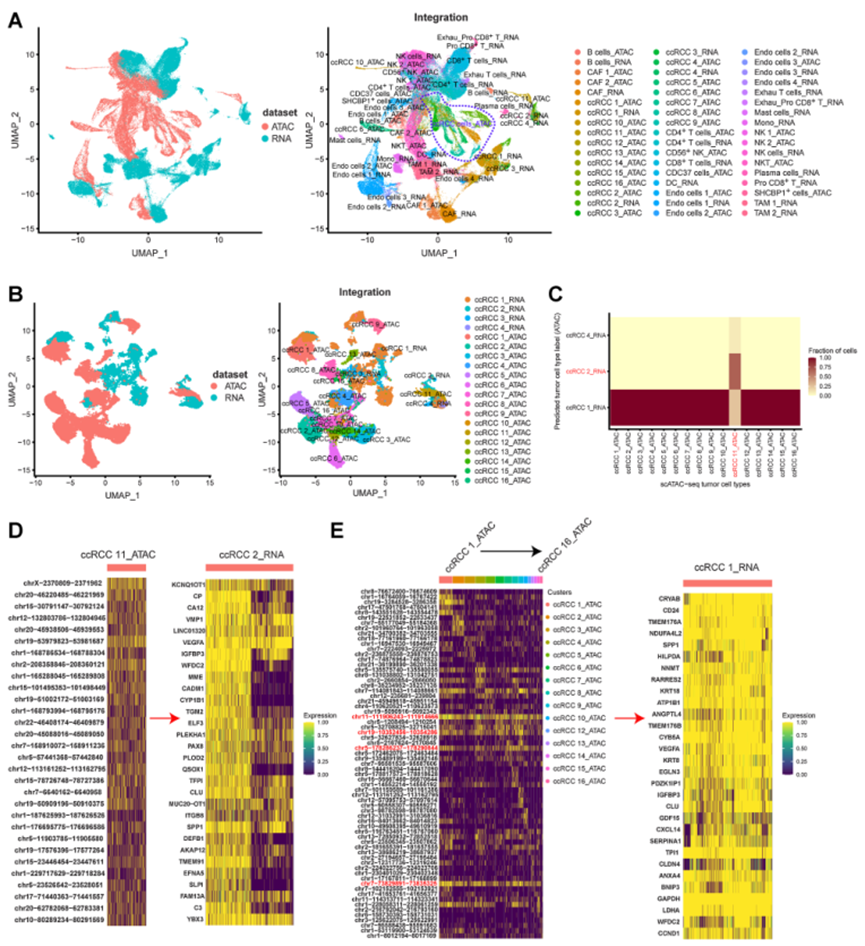
圖8 整合scRNA-seq和scATAC-seq分析
實驗方法:
臨床樣本收集,細胞培養,scATAC-seq,scRNA-seq,生物信息學分析, 核/質分離和qRT-PCR,流式細胞術,劃痕實驗,transwell實驗,RNA pull down+質譜,免疫印記
參考文獻:
Yu Z, Lv Y, Su C, Lu W, Zhang R, Li J, Guo B, Yan H, Liu D, Yang Z, Mi H, Mo L, Guo Y, Feng W, Xu H, Peng W, Cheng J, Nan A, Mo Z. Integrative Single-Cell Analysis Reveals Transcriptional and Epigenetic Regulatory Features of Clear Cell Renal Cell Carcinoma. Cancer Res. 2023 Mar 2;83(5):700-719. doi: 10.1158/0008-5472.CAN-22-2224. PMID: 36607615; PMCID: PMC9978887.






