簡單易行的微生物組和代謝組聯合分析
為了深入了解腸易激綜合征(IBS)中復雜的調控軸微生物-腸道-大腦,必須結合幾種生物學和臨床數據進行分析。本研究旨在識別與腸易激綜合征相關的糞便微生物群和代謝物,并描述腸易激綜合征的特定表型。本研究納入142名IBS患者和120名健康對照者(HC)的數據集,這些數據集具有廣泛的臨床、生物學和表型信息;使用質子核磁共振(1H-NMR)光譜和鳥槍法宏基因組測序(HG)測定糞便代謝物和腸道微生物組。本研究于2022年1月發表在《Gut Microbes》IF:10.245期刊上。
技術路線:
主要研究結果:
1、樣本基礎信息
糞便樣本來自314個(181例IBS患者,133例HC對照)受試者,受試者的基線信息如表1所示。糞便水1H-NMR光譜代謝分析數據采集自267例受試者,并配對262例(142例IBS患者,120例HC對照)的1H-NMR數據和糞便MGS數據,基線數據和總體數據間無差異。IBS組和HC組比較,作為腸腦交互作用改變的指標,IBS患者組的焦慮和抑郁得分較高,生活質量和高應激反應性等心理成分得分較低(表1),其余各項病理指標也顯著改變。

2、IBS患者和HC的糞便微生物組和代謝組數據的差異
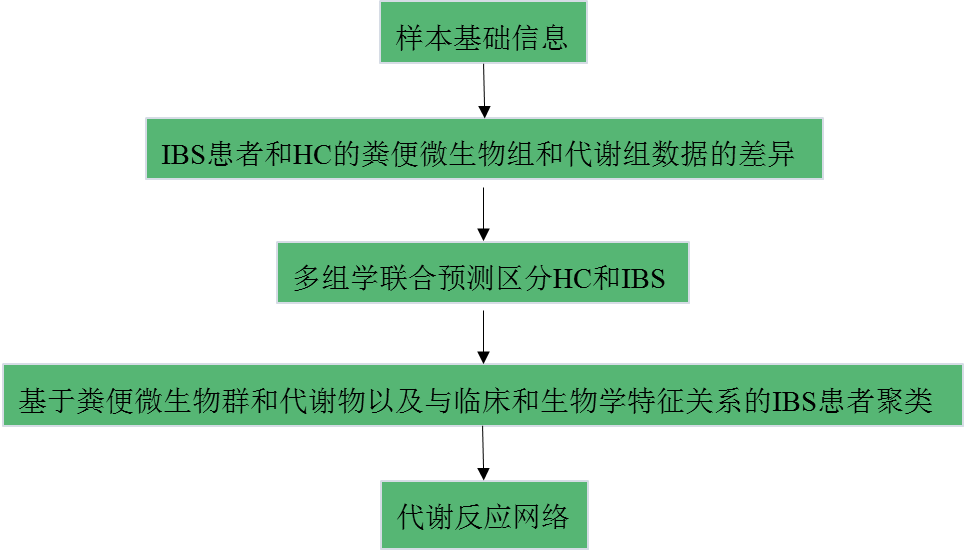
與HC組比較,辛普森的均勻度指標顯示IBS患者的腸道微生物α多樣性顯著降低(圖1a-1b)。當從不同的分類等級來區分HC和IBS患者時,兩組之間最好的區分不是在種水平,而是在科水平(圖1c)。在科水平,通過使用遞歸特征消除(RFE),HC和IBS患者之間的區分可以進一步提高到77.3%(AUC)(圖1d)。導致HC和IBS患者這種差異區分的細菌主要是圖1e所示的菌群。在1H-NMR光譜數據中,在HC和IBS患者間鑒定到50個特異性代謝物,基于這些代謝物,HC和IBS患者的區分可可達79.5%(AUC)(圖1f)。導致HC和IBS患者這種差異區分的代謝物如圖1g所示,其中10種代謝物在HC中富集,16種代謝物在IBS中高度富集。

圖1利用不同分類等級的微生物組和糞便水代謝物區分HC和IBS
3、多組學聯合預測區分HC和IBS
當聯合科水平的微生物群和糞便代謝物共同分析時,IBS和HC的區分可高達83.6%(AUC)(圖2a)。該模型由區分 HC 和 IBS 患者的微生物家族和代謝物組成(圖2b)。如圖2c所示,與單獨使用微生物群或代謝物區分HC和IBS比較,多組學聯合可以提供更好的區分結果。
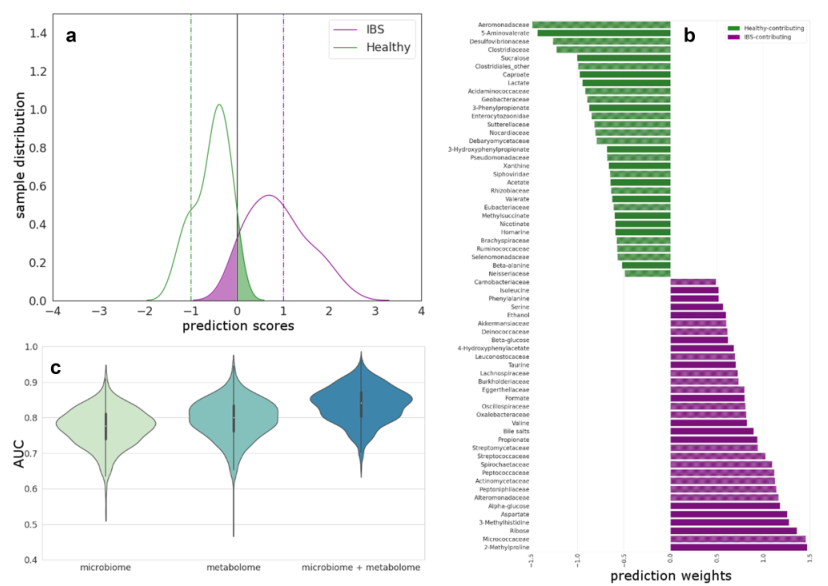
圖2多組學整合預測HC與IBS的差異
4、基于糞便微生物群和代謝物以及與臨床和生物學特征關系的IBS患者聚類
當只考慮IBS患者時,可以分別根據糞便微生物群和代謝物,并結合兩者的組學數據來識別不同的患者群(圖3a-3b)。對這些聚類進行分析,以了解該隊列中廣泛的臨床和生物學元數據的差異(圖3a),在多次檢測后發現,這些差異包括腸易激綜合征癥狀持續時間、應激引發的胃腸道癥狀、應激事件后腸易激綜合征的發生、血漿5-羥色胺(5-羥色胺)和5-羥吲哚乙酸、總膳食能量攝入和腹部手術史(圖3b)。
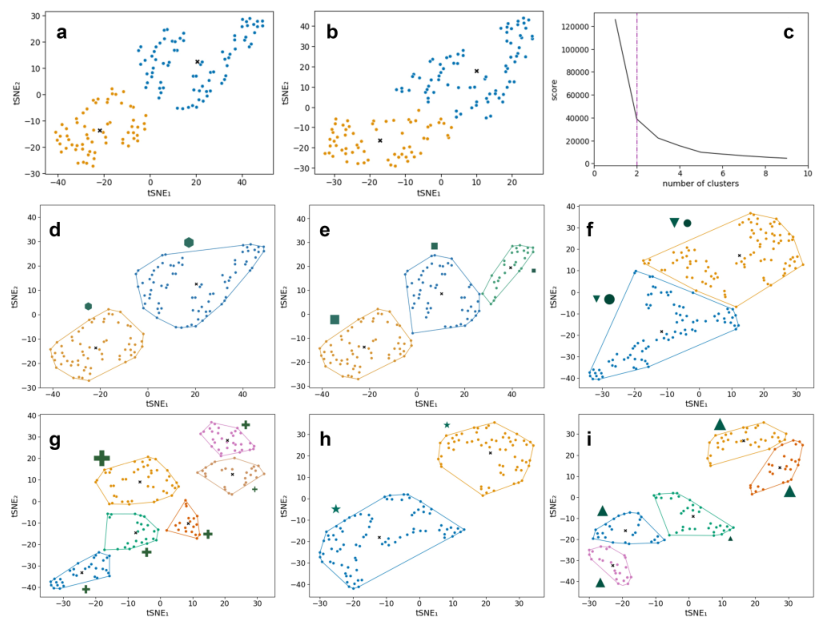
圖3a基于核t-SNE的無監督聚類
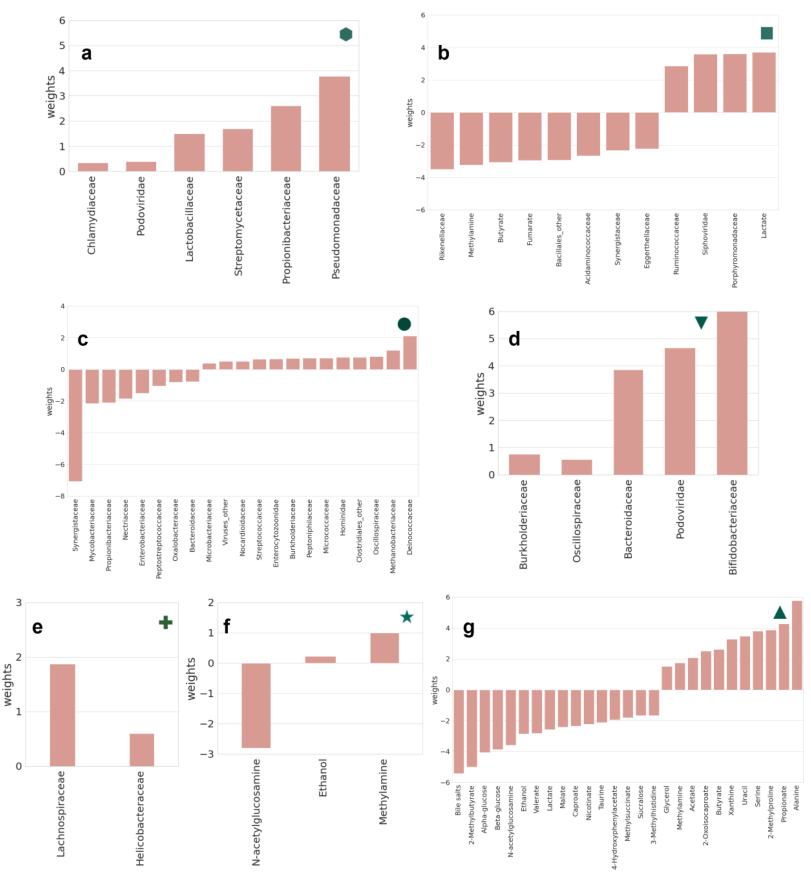
圖3b 臨床標志物SVR/SVC的模型重要特征在聚類間有顯著差異
5、代謝反應網絡
為了識別腸道菌群和相應代謝物之間的聯系,一個廣泛的代謝反應網絡被構建,其涉及IBS患者中顯著增加和降低的腸道菌群和糞便代謝物(圖4a)。在這幅交互圖中,左側顯示的是IBS(紫色)和HC(綠色)中增加的微生物家族;通過中心的酶,連接右側的糞便水代謝物,右側是IBS(紫色)和HC(綠色)中增加的代謝物。可以從此圖推斷糖分解和蛋白水解代謝活性的途徑。第一,丙酸輔酶A轉移酶,一種參與脂肪酸合成和氧化的微生物酶,存在于 14 個 IBS 和 13 個 HC 相關細菌家族中,可以產生乙酸鹽和乳酸(都是HC較高,圖4b)。輔酶A結合形式的乙酸或乳酸被釋放,輔酶A結合形式的丙酸由游離丙酸形成(IBS中較高)。第二,從酰基天冬氨酸底物產生天冬氨酸和羧酸鹽的一類普通天冬氨酸酰基酶(圖4c)。羧酸代謝物甲酸和乙酸可以由該酶介導的反應產生。IBS患者糞便中天門冬氨酸和甲酸含量較高,而HC中乙酸含量較高。第三,在5個HC和3個IBS相關微生物家族中發現的丙氨酸-乳酸連接酶,使用丙氨酸和乳酸作為底物。然而,IBS中丙氨酸的濃度更高,HC中乳酸的濃度更高。許多酶催化涉及多種代謝物的反應,這些反應都與IBS或HC有關。最后,肌肽合酶從底物-丙氨酸和3-甲基組氨酸產生鵝絲氨酸(圖4d)而beta-丙氨酸-組氨酸二肽酶則催化逆反應(圖4e)。
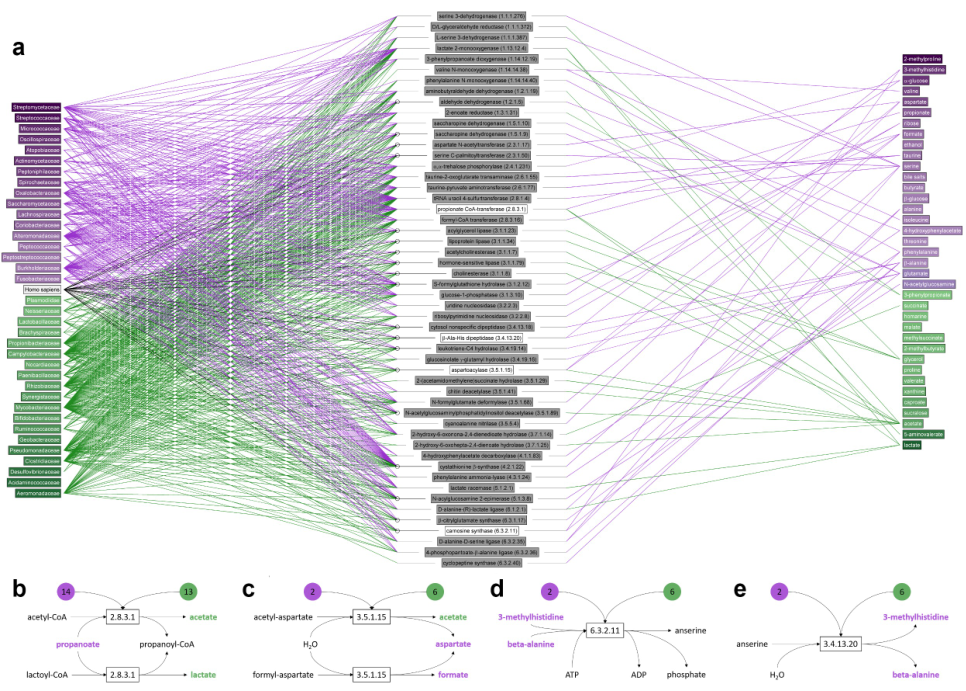
圖4在一個或多個微生物家族中發現的酶介導反應的代謝物
本研究已確定 IBS 的糞便微生物組-代謝組特征,這與改變的血清素代謝和與胃腸道癥狀相關的不利應激反應有關。與微生物組信息整合的代謝反應網絡顯示了宿主-微生物組相互作用的途徑。這些結果支持 IBS 發病機制中的微生物-腸-腦聯系。
參考文獻:
Mujagic Zlatan., Kasapi Melpomeni., Jonkers Daisy Mae., Garcia-Perez Isabel., Vork Lisa., Weerts Zsa Zsa R M., Serrano-Contreras Jose Ivan., Zhernakova Alexandra., Kurilshikov Alexander., Scotcher Jamie., Holmes Elaine., Wijmenga Cisca., Keszthelyi Daniel., Nicholson Jeremy K., Posma Joram M., Masclee Ad Am.(2022). Integrated fecal microbiome-metabolome signatures reflect stress and serotonin metabolism in irritable bowel syndrome. Gut Microbes, 14(1), 2063016. doi:10.1080/19490976.2022.2063016






